CUT&RUN 概述
了解 CUT&RUN —— ChIP-qPCR 和 ChIP-seq 的一种低细胞量备选方案,可用来分析染色质中的蛋白-DNA 相互作用。
什么是 CUT&RUN?
染色质免疫沉淀法 (ChIP) 和 ChIP-seq 等能够比对蛋白-DNA 相互作用的方法的开发,使得人们越来越了解到异常的表观遗传调节可以引起许多人类疾病。核酸酶靶向切割和释放 (CUT&RUN) 是一项可用于染色质分析的新技术。
它是如何工作的
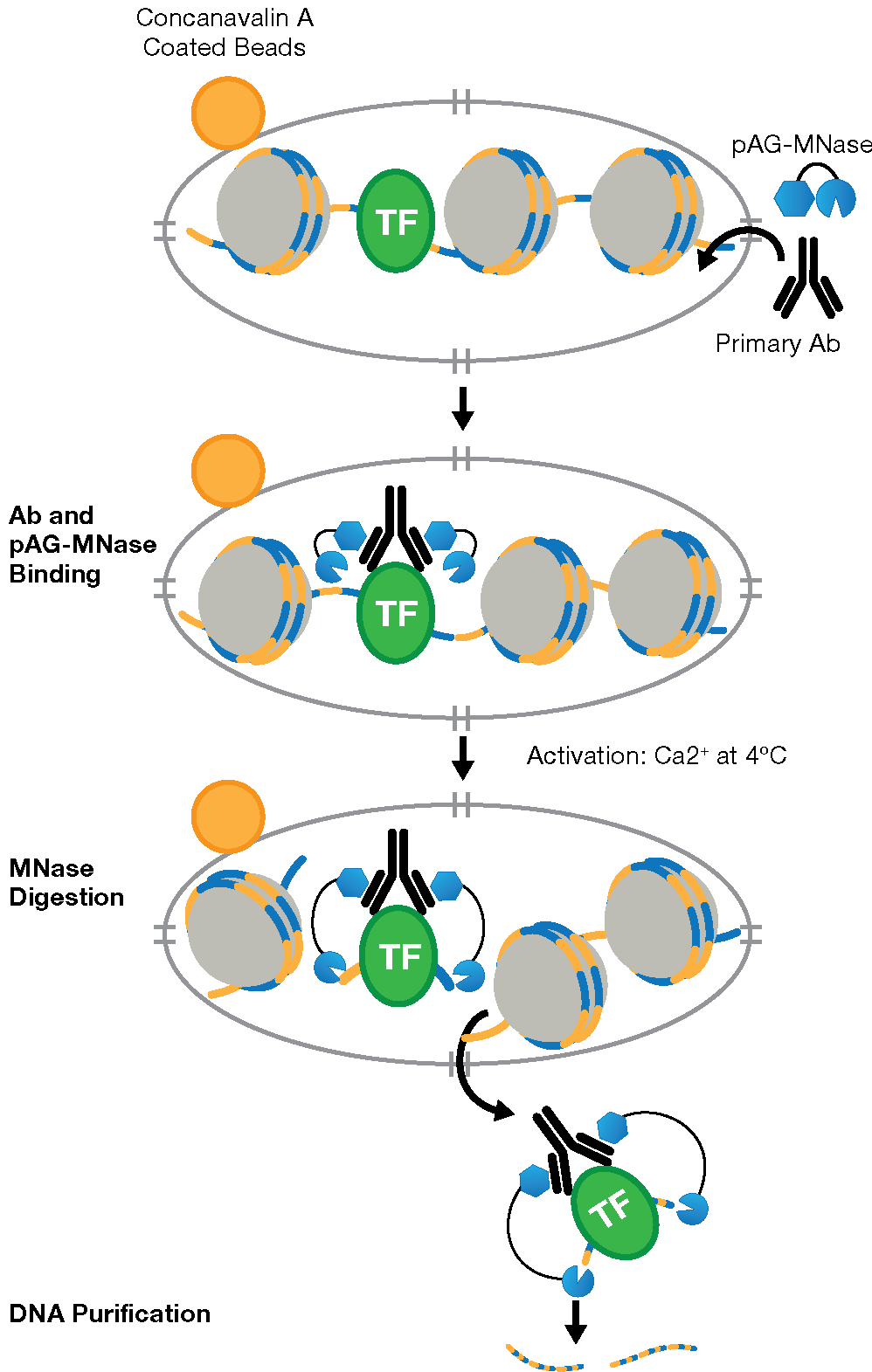
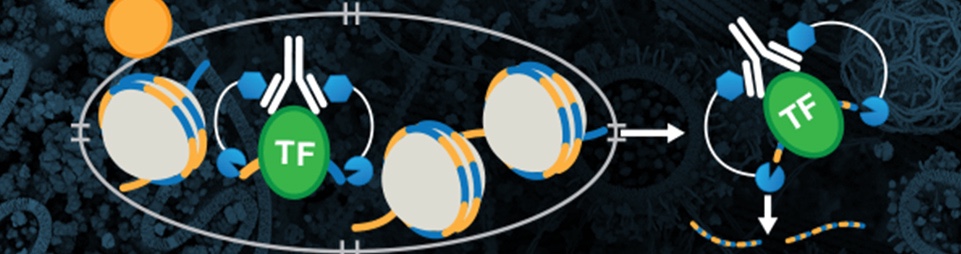
CUT&RUN 是一种使用靶标特异性一抗和 Protein A-Protein G-Micrococcal Nuclease (pAG-MNase) 分离特异性蛋白-DNA 复合体的体内方法1,2,3。从细胞到 DNA 只需 1 至 2 天,并且它可以自动化,实现最大通量和可重复性4。
为了分离目标蛋白-DNA 复合体,首先收集细胞,然后让其结合 Concanavalin A 包被的磁珠,以方便细胞处理,并在后续清洗过程中最大程度减少细胞丢失。细胞膜用洋地黄皂苷透化,便于一抗进入胞核,在胞核,一抗会结合组蛋白、转录因子或目标辅因子。pAG-MNase 融合蛋白的 pAG 结构域随后结合一抗重链,从而使酶靶向至目标染色质区域。添加 Ca2+ 可激活 pAG-MNase,以启动 DNA 消化。这会使被切割的染色质复合体从基因组染色质中扩散出来、脱离胞核并进入样品上清液,使用 DNA 离心柱或苯酚/氯仿提取后再行乙醇沉淀的方法便可从上清液中收集这些被切割的染色质复合体。
纯化、富集的 DNA 随后使用 qPCR 检测和定量,或用下一代测序分析 (NGS) 与全基因组比对构建DNA 测序文库。
CUT&RUN 的优点
Cell Signaling Technology® (CST) 提供的 CUT&RUN 试剂盒可克服其他全基因组比对技术面临的许多挑战。CST 提供的 CUT&RUN 试剂盒的优点包括:
结果立等可取 | 从细胞到 DNA 需 1 到 2 天 |
样品量要求少 | 推荐 100000 个细胞,如果需要,验证 5-20000 细胞(取决于靶标类型) |
节省测序成本 | 仅需 300-500 万个高质量读长 |
避免出现“交联”假象 | 一种使用天然染色质进行的体内方法 |
抗体通用 | 兼容于兔抗体和小鼠抗体 |
靶标通用 | 针对组蛋白、组蛋白修饰、转录因子和辅因子生成测序和/或 qPCR 数据 |
结果可重复 | 加入对照 DNA 可标准化样品间的信号 |
所需样品少
仅需培养 100,000 个细胞即可观察蛋白-DNA 相互作用和/或得到有关低丰度细胞类型的表观遗传信息,节省时间。
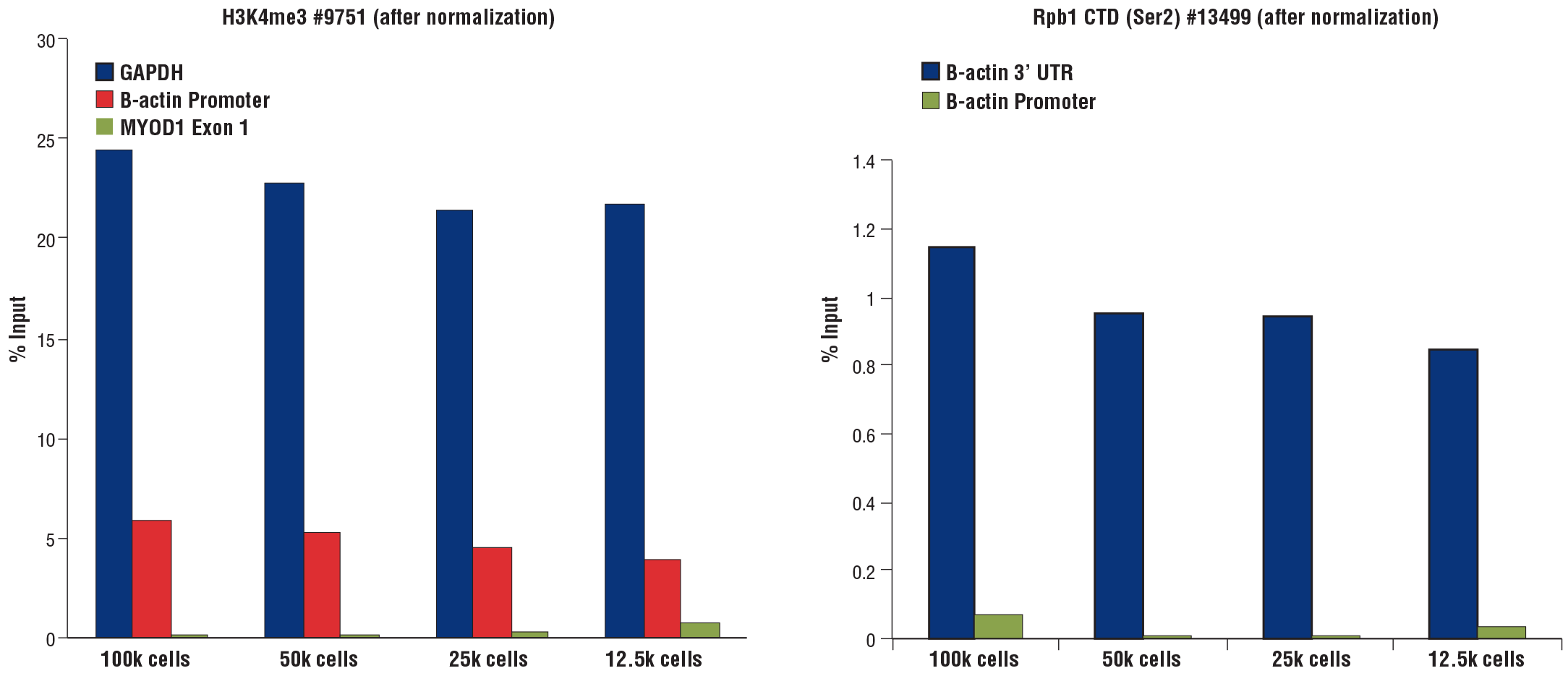
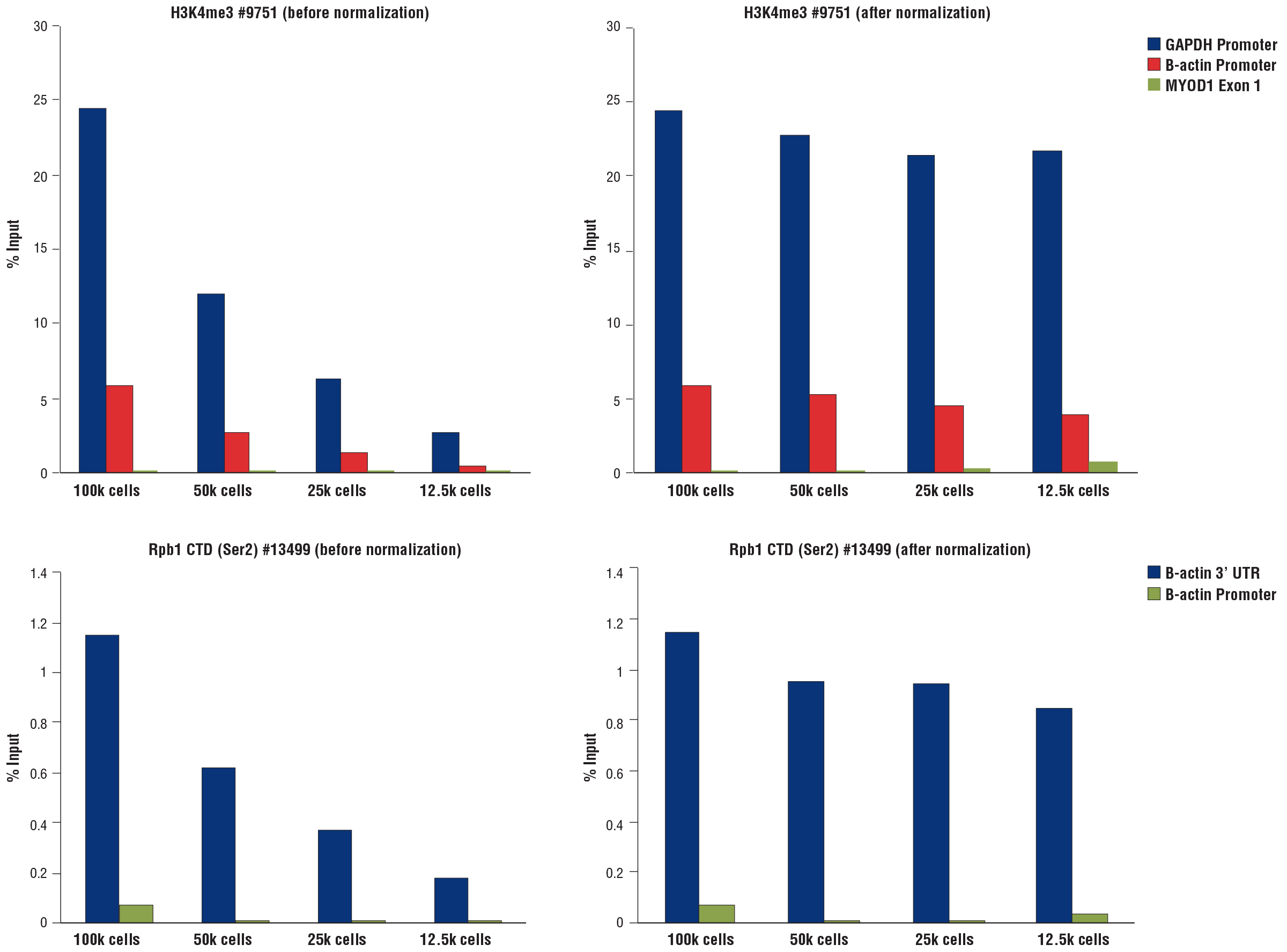
使用更少数量的 HCT 116 细胞来进行使用 CUT&RUN Assay Kit #86652 的 CUT&RUN 时,获得了相似的结果。使用 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751(左小图)或 Phospho-Rpb1 CTD (Ser2) (E1Z3G) Rabbit mAb #13499(右小图)进行测定。使用 SimpleChIP® Universal qPCR Master Mix #88989 以及 SimpleChIP® Human GAPDH Exon 1 Primers #5516、SimpleChIP® Human β-Actin Promoter Primers #13653、SimpleChIP® Human β-Actin 3' UTR Primers #13669 和 SimpleChIP® Human MyoD1 Exon 1 Primers #4490 进行实时 PCR 来对富集的 DNA 进行定量。每份样品中的免疫沉淀 DNA 数量表示为与输入染色质总量(100,000 个细胞的输入百分比)相对应的信号。使用加入到每次反应中的 spike-in DNA 来标准化数据。
快速得到结果
节省富集靶标染色质所花费的时间。从细胞到 DNA 仅需 1 到 2 天。
节省测序成本
背景相对较低是指您可在测序深度较低的情况下,区分您的信号与基因组背景噪音。仅需 300 - 500 万次高质量读长便可获得测序数据,显著降低了您的测序成本。有额外收获吗? 这种方法的低背景还可能用于分析低信号基因组特征。
靶标通用
纯化、富集的 DNA 可用于测序和/或 qPCR 实验,以研究组蛋白、组蛋白修饰、转录因子和辅因子。
组蛋白修饰
在研究组蛋白修饰时,CUT&RUN Assay Kit #86652 可达到与 ChIP-qPCR 和 ChIP-seq 相同的效果,但仅需 100,000 个细胞。
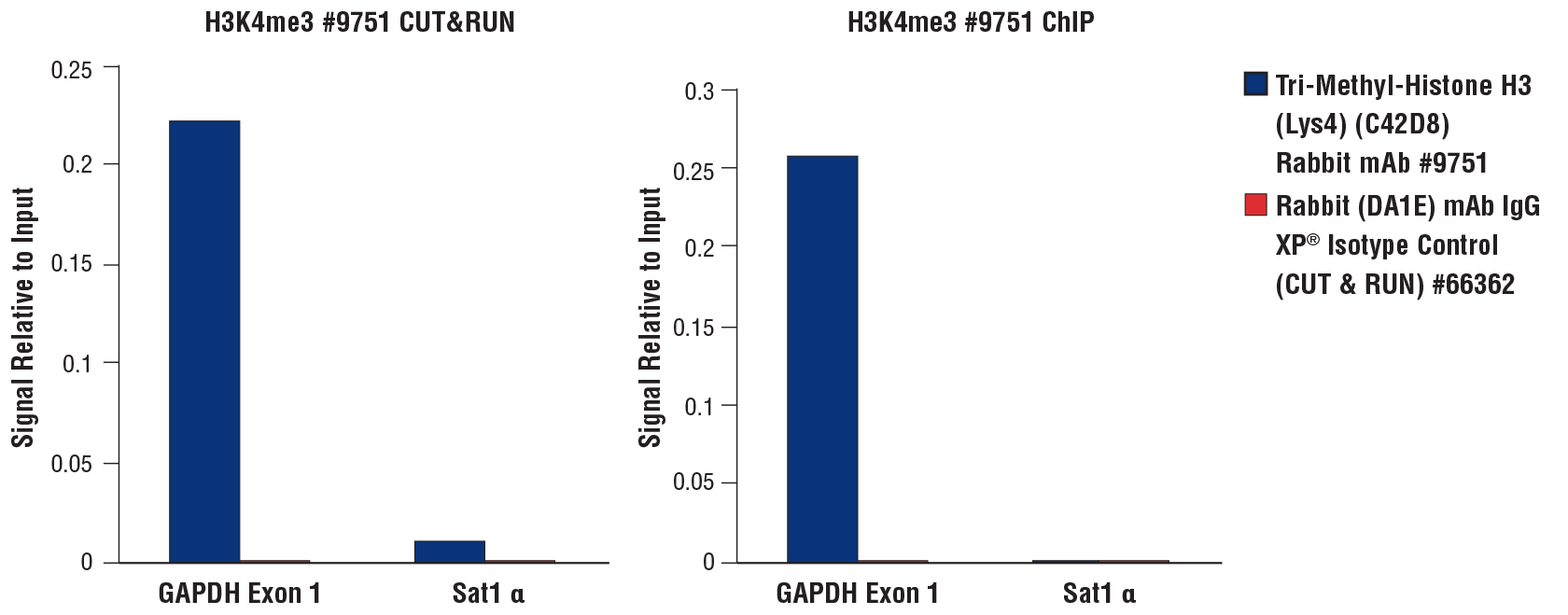
通过使用 CUT&RUN Assay Kit #86652(左小图)或 SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005(右小图),用 HCT 116 细胞(CUT&RUN 用 1x10^5 个细胞,ChIP 用 4x10^6 个细胞)与 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 或 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362 进行 CUT&RUN 和 ChIP 实验。使用 SimpleChIP® Universal qPCR Master Mix #88989 以及 SimpleChIP® Human GAPDH Exon 1 Primers #5516 和 SimpleChIP® Human α Satellite Repeat Primers #4486 进行实时 PCR 来对富集的 DNA 进行定量。每份样品中的免疫沉淀 DNA 数量表示为与输入染色质总量(相当于 1)相对应的信号。
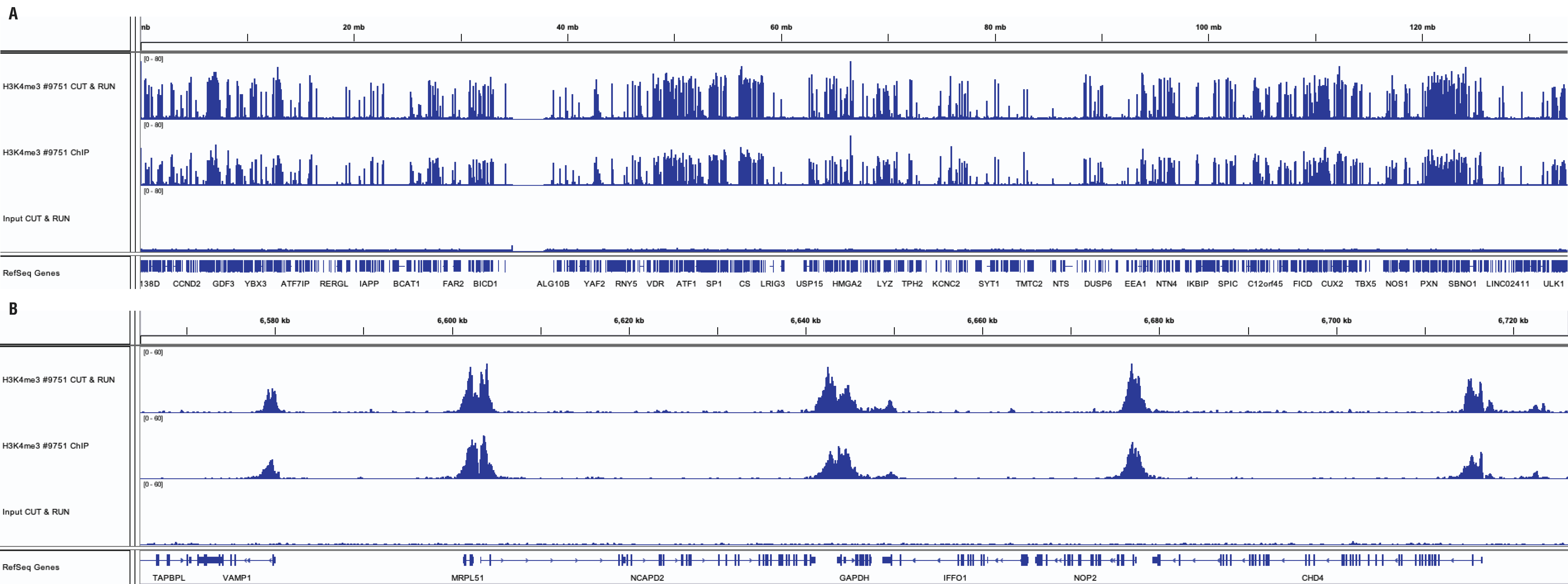
通过使用 CUT&RUN Assay Kit #86652 或SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005,用 HCT 116 细胞(CUT&RUN 用 1×10^5 个细胞,ChIP 用 4×10^6 个细胞)和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 进行 CUT&RUN 和 ChIP 实验。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。小图 A 比较了 H3K4me3 在染色体 12 上的富集,而小图 B 则比较了 H3K4me3 在已知靶标 GAPDH 基因上的富集。input通道为 CUT&RUN 中的input样品。
转录因子
在研究转录因子 DNA-蛋白相互作用时,CUT&RUN Assay Kit #86652 可达到与 ChIP-qPCR 和 ChIP-seq 相同的效果,但仅需 100,000 个细胞。
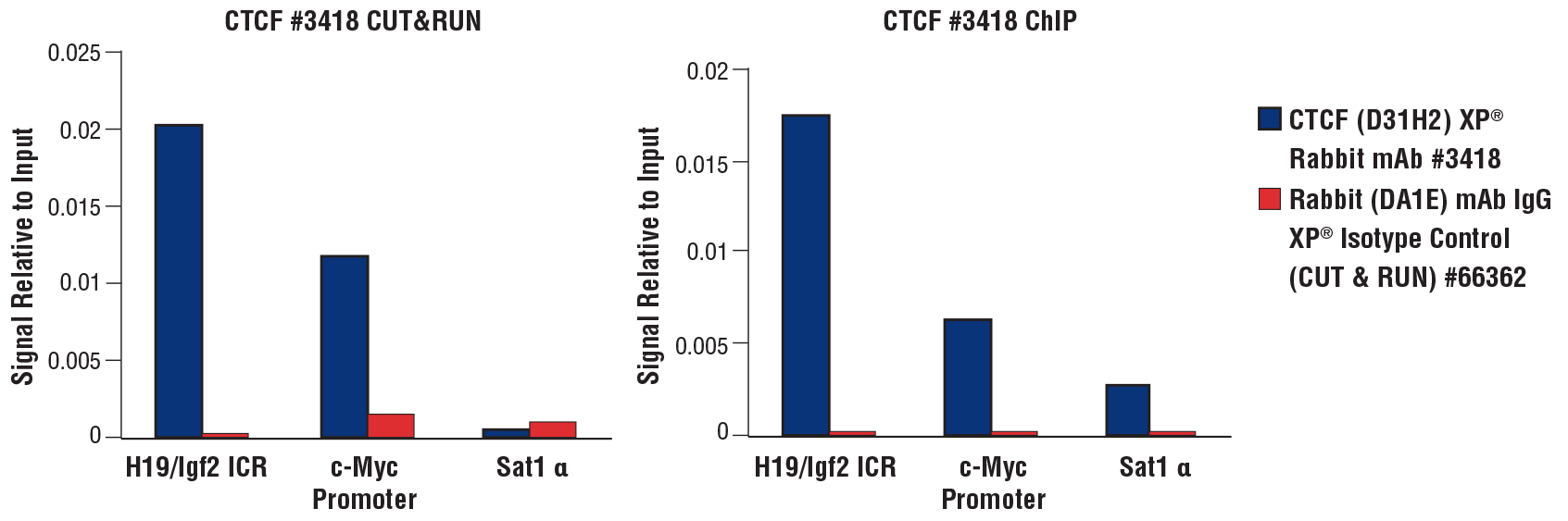
通过使用 CUT&RUN Assay Kit #86652(左小图)或 SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005(右小图),用 HCT 116 细胞(CUT&RUN 用 1×10^5 个细胞,ChIP 用 4×10^6 个细胞)与 CTCF (D31H2) XP® Rabbit mAb #3418 或 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362 进行 CUT&RUN 和 ChIP 实验。使用 SimpleChIP® Universal qPCR Master Mix #88989 以及 human c-Myc promoter primers、SimpleChIP® Human H19/Igf2 ICR Primers #5172 和 SimpleChIP® Human α Satellite Repeat Primers #4486 进行实时 PCR 来对富集的 DNA 进行定量。每份样品中的免疫沉淀 DNA 数量表示为与输入染色质总量(相当于 1)相对应的信号。
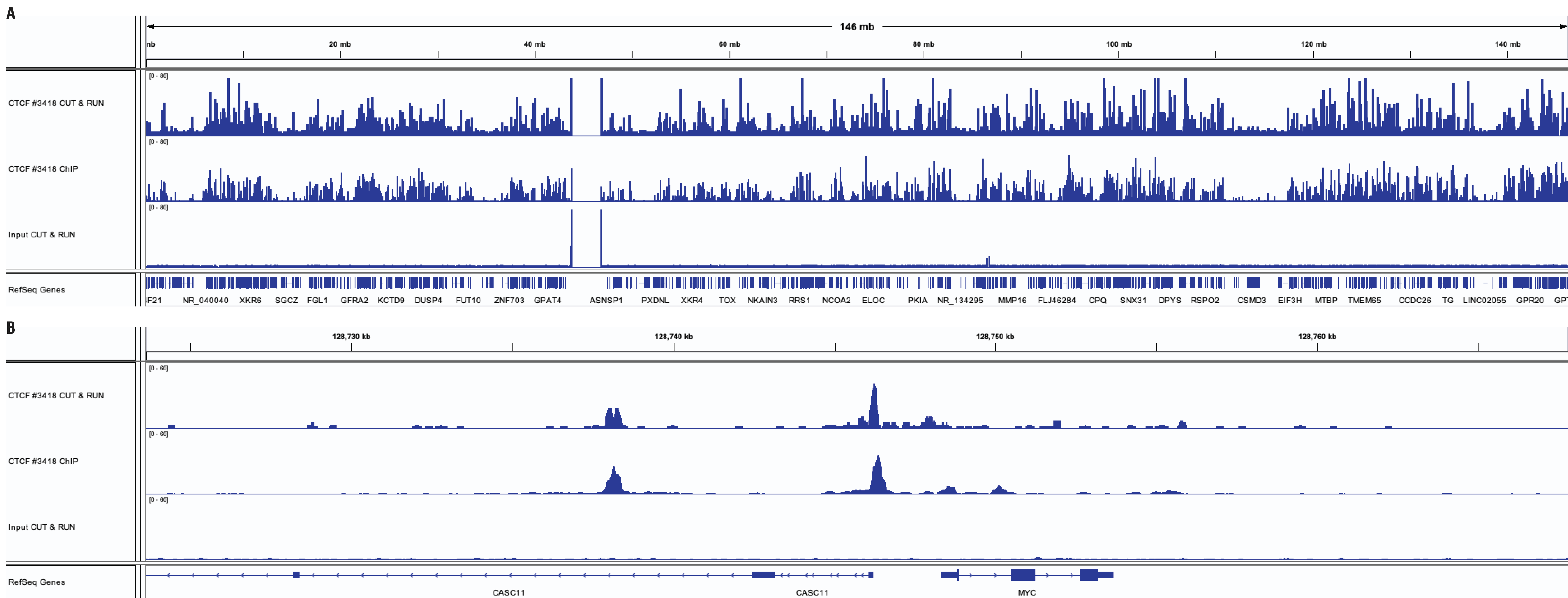
通过使用 CUT&RUN Assay Kit #86652 或 SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005,用 HCT 116 细胞(CUT&RUN 用 1×10^5 个细胞,ChIP 用 4x10^6 个细胞)和 CTCF (D31H2) XP® Rabbit mAb #3418 进行 CUT&RUN 和 ChIP 实验。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。小图 A 比较了 CTCF 在 8 号染色体上的富集,而小图 B 则比较了 CTCF 在其已知靶标 MYC 基因上的富集。input通道为 CUT&RUN 中的input样品。
辅因子
在研究辅因子 DNA-蛋白相互作用时,CUT&RUN Assay Kit #86652 可达到与 ChIP-qPCR 和 ChIP-seq 相同的效果,但仅需 100,000 个细胞。
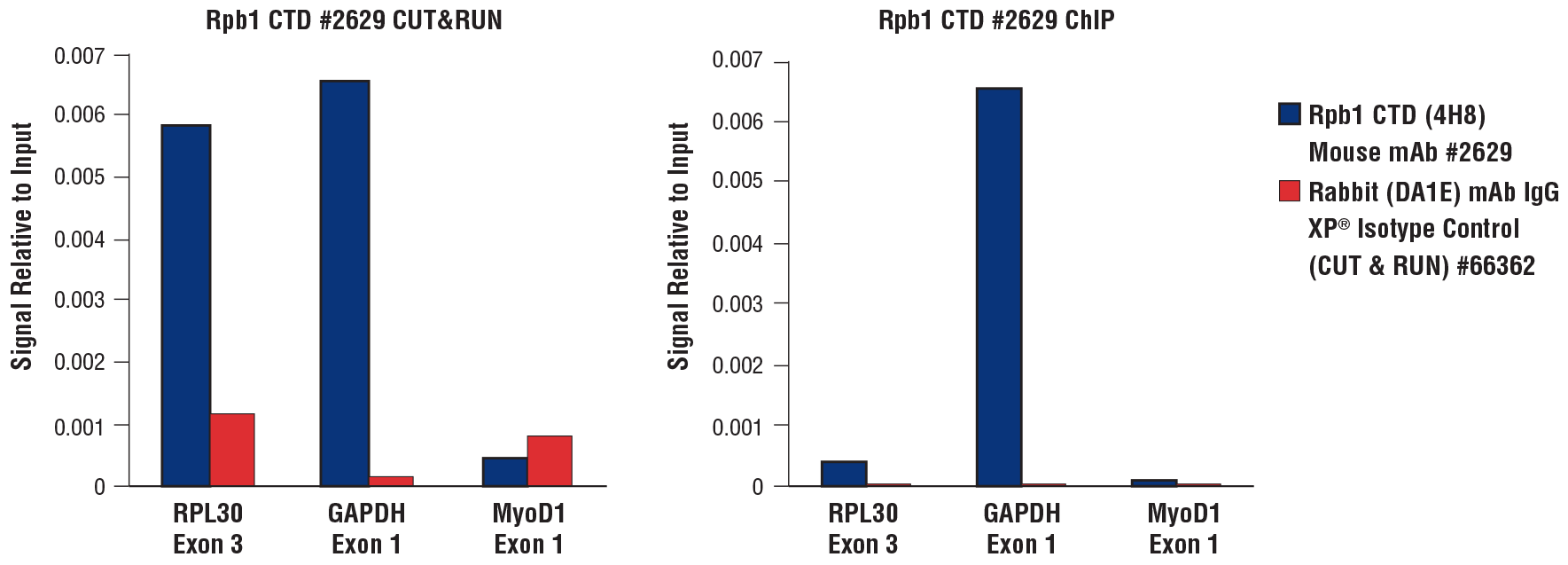
通过使用 CUT&RUN Assay Kit #86652(左小图)或 SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005(右小图),用 HeLa 细胞(CUT&RUN 用 1×10^5 个细胞,ChIP 用 4×10^6 个细胞)与 Rpb1 CTD (4H8) Mouse mAb #2629 或 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362 进行 CUT&RUN 和 ChIP 实验。使用 SimpleChIP® Universal qPCR Master Mix #88989 以及 SimpleChIP® Human RPL30 Exon 3 Primers #7014、SimpleChIP® Human GAPDH Exon 1 Primers #5516 和 SimpleChIP® Human MyoD1 Exon 1 Primers #4490 进行实时 PCR 来对富集的 DNA 进行定量。每份样品中的免疫沉淀 DNA 数量表示为与输入染色质总量(相当于 1)相对应的信号。
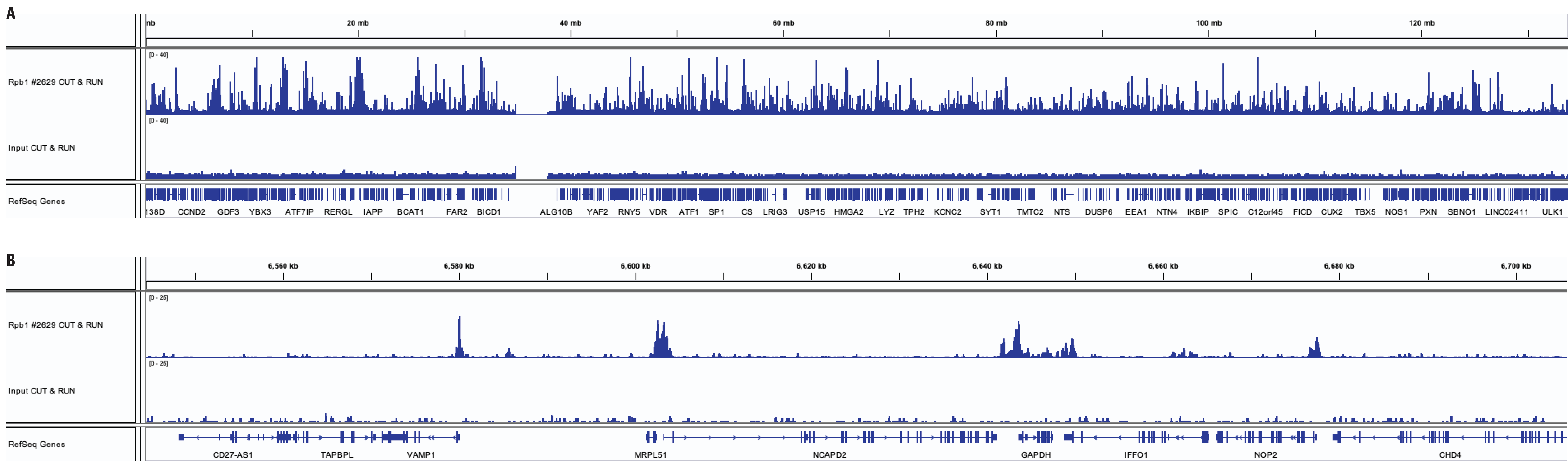
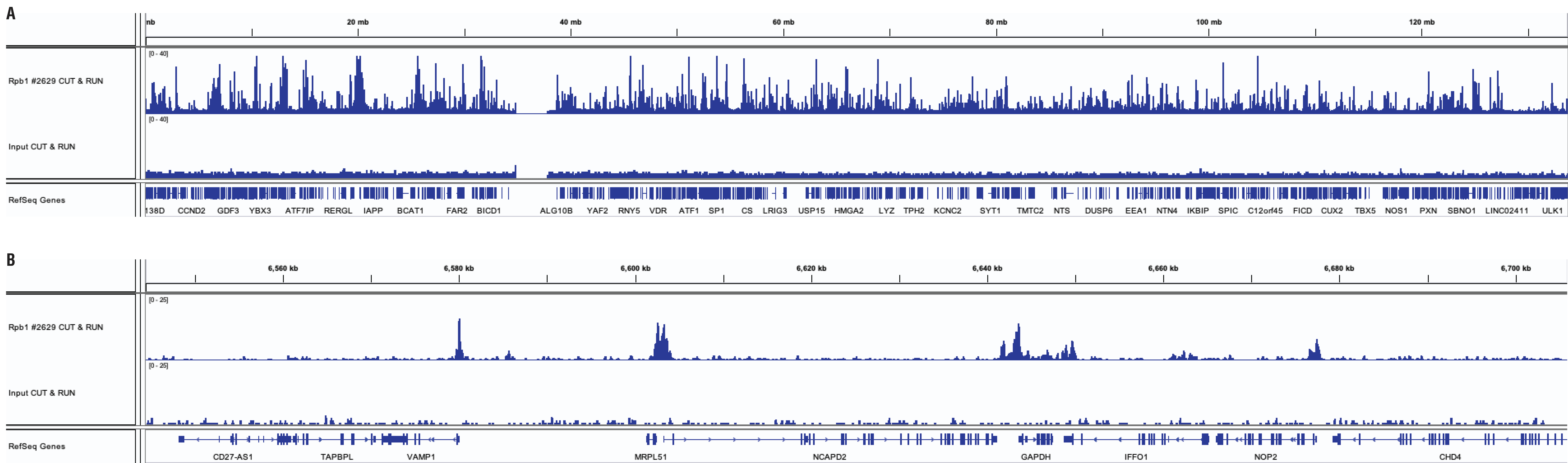
通过使用 CUT&RUN Assay Kit #86652,用 HeLa 细胞(CUT&RUN 用 1x10^5 个细胞,ChIP 用 4×10^6 个细胞)和 Rpb1 CTD (4H8) Mouse mAb #2629 进行 CUT&RUN 和 ChIP 实验。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。小图 A 比较了 Rpb1 在染色体 12 上的富集,而小图 B 则比较了 Rbp1 在已知靶标 GAPDH 基因上的富集。input通道为 CUT&RUN 中的input样品。
抗体范围广
pAG-MNase 结合鼠抗和兔抗,从而增加了适用于 CUT&RUN 检测的抗体目录。
兔抗
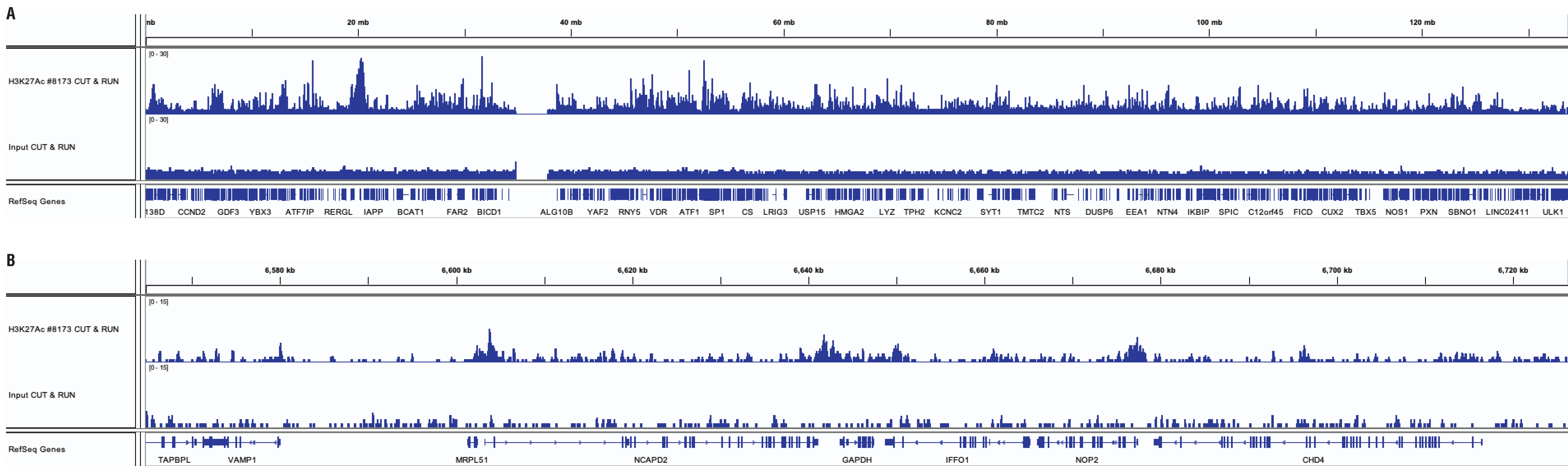
通过使用 CUT&RUN Assay Kit #86652,用 HeLa 细胞和 Acetyl-Histone H3 (Lys27) (D5E4) XP® Rabbit mAb #8173 进行 CUT&RUN 和 ChIP 实验。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。小图 A 比较了 H3K27Ac 在 12 号染色体上的富集,而小图 B 则比较了 H3K27Ac 在其已知靶标 GAPDH 基因上的富集。
小鼠抗体
通过使用 CUT&RUN Assay Kit #86652,用 HeLa 细胞和 Rpb1 CTD (4H8) Mouse mAb #2629 进行 CUT&RUN 和 ChIP 实验。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。小图 A 比较了 Rpb1 在染色体 12 上的富集,而小图 B 则比较了 Rbp1 在已知靶标 GAPDH 基因上的富集。
可重复的结果
用添加异源 DNA 标准化您的数据,即可提升您的实验可重复性。对某个指定实验内或不同实验之间的样品进行标准化
使用因 qPCR 分析而加入的 DNA 对 CUT&RUN 检测信号进行标准化。使用 CUT&RUN Assay Kit #86652、数量减少的 HCT 116 细胞与 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751(顶部小图)或 Phospho-Rpb1 CTD (Ser2) (E1Z3G) Rabbit mAb #13499(底部小图)进行 CUT&RUN 测定。使用 SimpleChIP® Universal qPCR Master Mix #88989 以及 SimpleChIP® Human GAPDH Exon 1 Primers #5516、SimpleChIP® Human β-Actin Promoter Primers #13653、SimpleChIP® Human β-Actin 3' UTR Primers #13669 和 SimpleChIP® Human MyoD1 Exon 1 Primers #4490 进行实时 PCR 来对富集的 DNA 进行定量。每份样品中的免疫沉淀 DNA 数量表示为与输入染色质总量(100,000 个细胞的输入百分比)相对应的信号。左图中为非标准化的富集结果。在每次反应中,按照与起始细胞数量成比例的方式添加 Sample Normalization Spike-In DNA。根据每份样品中添加 DNA 所产生的 qPCR 信号差异,将 CUT&RUN 信号标准化为含 100,000 个细胞的样品。右图显示了标准化后的富集。
避免出现“交联假象”
CUT&RUN 适用于天然染色质,并且无需进行可能会导致“交联假象”的交联或免疫沉淀。
产品供应
无论您是一个才刚开始在研究中探索表观遗传学的新手,还是一位专家,CST 都可为您的 CUT&RUN 检测提供灵活的解决方案。您所需要提供的就是针对您的目的蛋白的一抗。CST 可提供订购便捷 的CUT&RUN 试剂盒中的所有其他用品,包括您所需要的缓冲液和试剂,以及详细的实验步骤。如果您愿意,也可仅订购 pAG-MNase 和spike-in DNA。
像所有的 CST 产品一样,我们的 CUT&RUN 检测产品均在内部经过严格验证,以确保它们在您的实验中有效并产生您能够信任的数据。
了解我们如何验证和优化我们的 CUT&RUN 试剂盒以产生更好的信噪比,需要更少的细胞来分析蛋白质-DNA 相互作用,并与组织样本兼容。及时了解我们对 CUT&RUN 检测试剂盒的所有实验步骤改进。
货号 | 产品 |
|---|---|
| 86652 | CUT&RUN Assay Kit |
| 40366 | CUT&RUN pAG-MNase and Spike-in DNA |
| 14209 | DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN) |
| 88989 | SimpleChIP® Universal qPCR Master Mix |
| 56795 | DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 |
| 47538 | Multiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) |
| 29580 | Multiplex Oligos for Illumina (Single Index Primers) (ChIP-seq, CUT&RUN) |
| 66362 | Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) |
| 93569 | Concanavalin A Magnetic Beads and Activation Buffer |
| 16359 | Digitonin Solution |
| 27287 | 100X Spermidine |
| 7012 | Protease Inhibitor Cocktail (200X) |
| 7013 | RNAse A (10 mg/ml) |
| 10012 | Proteinase K (20mg/ml) |
| 31415 | CUT&RUN 10X Wash Buffer |
| 15338 | CUT&RUN Antibody Binding Buffer |
| 48105 | CUT&RUN 4X Stop Buffer |
| 42015 | CUT&RUN DNA Extraction Buffer |
| 20533 | 10% SDS Solution |
| 7005 | Glycine Solution (10X) |
| 12606 | 16% Formaldehyde Methanol Free |
| 9872 | PBS-1X pH 7.2 (Sterile) |
参考文献
- Skene PJ, et al. Targeted in situ genome-wide profiling with high efficiency for low cell numbers. (2018) Nat. Protoc 13(5), 1006-1019. Pubmed 29651053
- Meers MP, et al. Improved CUT&RUN chromatin profiling and analysis tools. (2019) BioRxiv 1, 569129. bioRxiv 569129
- Skene PJ, and Henikoff S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. (2017) Elife 6, e21865. Pubmed 28079019
- Janssens DH, et al. Automated in situ chromatin profiling efficiently resolves cell types and gene regulatory programs. (2018) Epigenetics Chromatin 22(1), 74. Pubmed 30577869